Dans la même rubrique
-
Partager cette page
Épidémies : utilisation d’approches moléculaires pour suivre et comprendre la circulation des virus dans des populations animales – des développements qui s’inscrivent dans l’approche "One Health"
Alors que des cas de grippe aviaire chez l’homme ont été récemment recensés et que la journée internationale de la préparation aux épidémies se profile, des chercheurs de l’Université libre de Bruxelles publient une nouvelle étude qui se base sur des approches moléculaires pour mesurer et comparer la capacité de dispersion de virus dans des populations animales.
Dans une étude publiée dans la revue PLoS Biology ce 3 décembre 2024, des chercheurs de l’Université libre de Bruxelles (ULB) et leurs collaborateurs exploitent la très grande variabilité génétique présente chez les virus pour mesurer leur capacité de dispersion. Les chercheurs ont évalué la performance de plusieurs statistiques de dispersion pour ensuite estimer et caractériser la vitesse de diffusion de près de 20 virus différents impactant des populations animales, allant du virus de la rage à celui de la grippe aviaire. « Grâce à la très grande vitesse évolutive des virus, les approches moléculaires permettent d’estimer à quel point deux cas d’infection sont plus ou moins éloignés au sein d’une chaine de transmission. Cette source d’information peut ensuite être exploitée pour mieux comprendre l’histoire de dispersion d’un virus et estimer sa vitesse de diffusion dans une population », explique l’épistémologiste Simon Dellicour (Faculté des Sciences), auteur de l’étude.
Les analyses révèlent une large gamme de vitesses de diffusion qui reflètent majoritairement la capacité de dispersion de l’espèce animale hôte principalement infectée mais aussi, dans certains cas, la probable signature de rapides événements de dispersion longue distance provoqués par des déplacements d'origine humaine comme le commerce d'animaux. C’est par exemple probablement le cas pour l’importante vitesse de diffusion estimée pour le virus Getah et le deltacoronavirus infectant tous deux la population de cochons domestiques en Chine. Parmi les jeux de données que les chercheurs ont analysés, la plus faible capacité de dispersion a logiquement été estimée pour des virus dont la vitesse de déplacement de l’espèce hôte est limitée. C’est par exemple le cas du Nova virus circulant au sein d’une population de taupes, ou encore le virus de Lassa circulant en Afrique chez une espèce de rongeur. A l’autre bout du spectre, la capacité de dispersion la plus élevée a été estimée pour le virus du Nil Occidental dont la propagation en Amérique du Nord a été rapidement conduite par des oiseaux infectés volant sur de longues distances.
L’étude s’inscrit dans l’approche « One Health » consistant à intégrer de manière systémique santés environnementale, animale et humaine. Si les chercheurs s’intéressent ici à des virus circulant principalement dans des populations animales, bon nombre de ces virus viennent aussi infecter l’homme, avec un impact considérable sur la santé publique. C’est par exemple le cas du virus de la rage qui, sans transmission d’homme à homme, tue aujourd’hui encore près de 60 000 personnes chaque année, et ce malgré la disponibilité de vaccins et d’un traitement post-morsure. « Cette maladie constitue un exemple démontrant que santés animale et humaine ne peuvent définitivement pas être dissociées », souligne Simon Dellicour. Dans un tel contexte, comprendre la dynamique de circulation du virus dans différents pays et configurations s’avère être une des pièces du puzzle pour configurer les stratégies d’intervention les plus efficaces possibles.
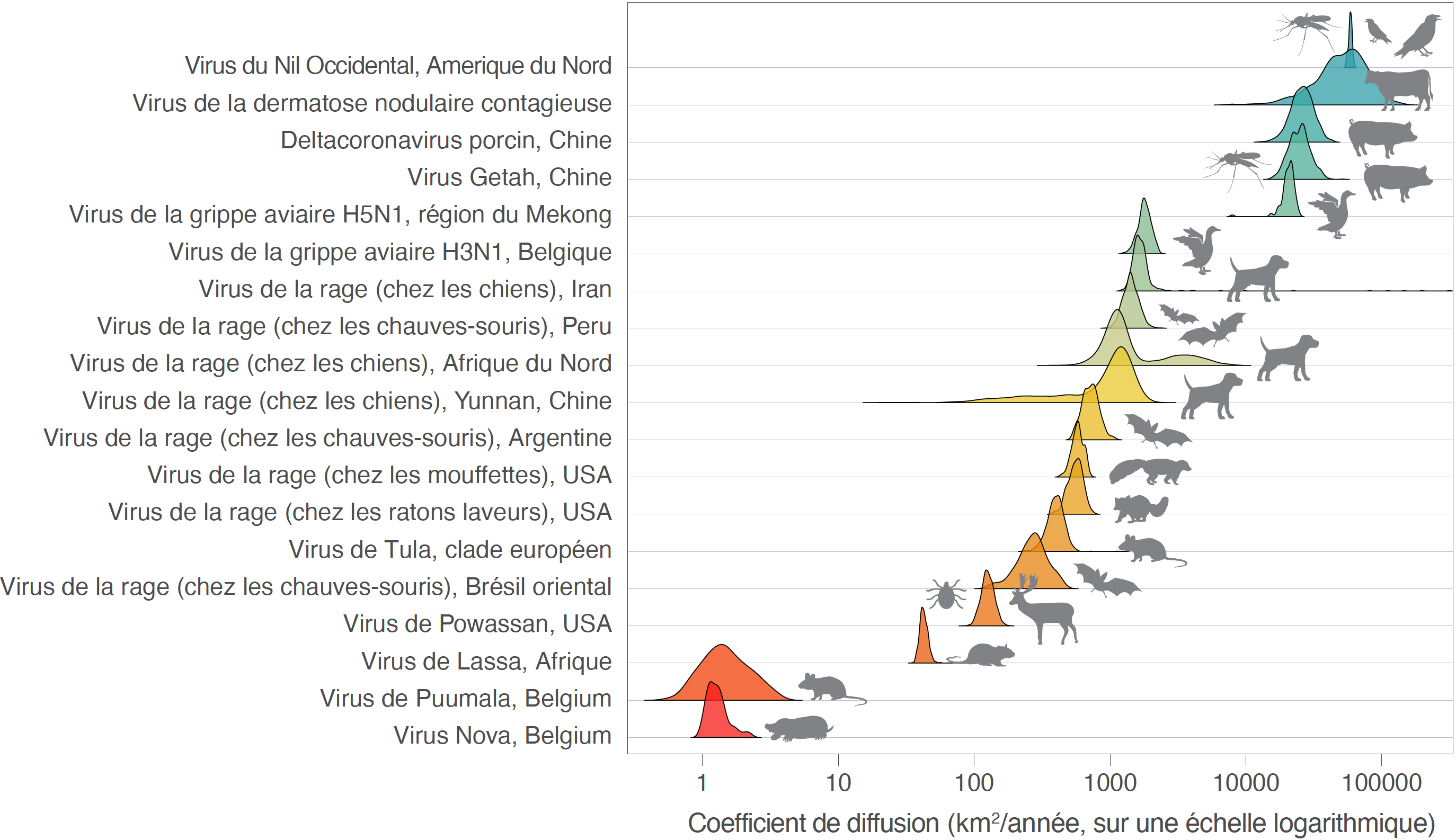
Illustration adaptée et traduite d’une des figures de l’étude : comparaison des capacités de dispersion estimées pour différents virus circulant dans des populations animales. Source : Dellicour et al. (2024, PLoS Biology).
Référence de l’étude : Dellicour S, Bastide P, Rocu P, Fargette D, Hardy OJ, Suchard MA, Guindon S, Lemey P (2024). How fast are viruses spreading in the wild? PLoS Biology 22: e3002914.
Lien vers l’étude : https://doi.org/10.1371/journal.pbio.3002914
Contact scientifique : Simon Dellicour, Faculté des Sciences.
E-mail : simon.dellicour@ulb.be|Téléphone : sur demande à presse@ulb.be
|
27 décembre 2024 |
Contactez-nous!
